 加载中...
加载中...
 日期:2022-09-30
日期:2022-09-30
 于举行份子表达操控中,常常听到或者使用到的一个技能即是Cre/Loxp或者Flp/FRT重组酶体系。简朴来讲,这种重组酶体系是经由过程特异位点重组酶(Site-specific recombinases, SSRs)介导重组酶特异辨认位点(Recombination tar-get sites, RTs)间的重组,来实现特异位点的基因敲除了、基因插入、基因翻转以及基因易位等操作。因为该技能可以或许有用降服其他类型重组技能的非特异性或者重组效率低等错误谬误,最近几年来已经逐渐于功效基因研究范畴盘踞了主导职位地方。 Cre-loxP的基来源根基理Cre是一种重组酶,来历在P1噬菌体,其基因编码区序列全长1029bp,为38kDa巨细的、由343个氨基酸构成的多肽单体卵白。Cre重组酶的C-结尾布局域包罗催化活性位点,可以或许催化DNA份子中特定位点之间的重组,同时,Cre还能辨认特异的DNA序列,即loxP位点,使两个loxP位点间发生基因重组。LoxP是Locus of X-overP1的缩写,是位在P1噬菌体中长度为34bp的一段序列,由两个13bp的反向回文序列以及8bp的中间距离序列配合构成,反向回文序列是Cre重组酶的辨认以及联合区域,距离序列决议LoxP序列的标的目的。 Cre/loxP引诱基因重组的体式格局一般而言,当细胞基因组内存于两个LoxP位点时,Cre重组酶会引诱两个LoxP位点间的序列发生重组。重组的成果取决在两个loxP位点的标的目的,重要有如下几种可能:① 假如两个LoxP位点位在一条DNA链上且标的目的不异,Cre重组酶能有用地删除了两个LoxP位点间的序列;② 假如两个LoxP位点位在一条DNA链上但标的目的相反,Cre重组酶能引诱两个LoxP位点间的序列翻转;③假如两个LoxP位点别离位在两条差别的DNA链或者染色体上,Cre重组酶能引诱两条DNA链发生互换或者染色体易位,即基因转座④假如四个loxP位点别离位在两条差别的DNA链或者染色体上,Cre重组酶能引诱loxP间的序列交换。
于举行份子表达操控中,常常听到或者使用到的一个技能即是Cre/Loxp或者Flp/FRT重组酶体系。简朴来讲,这种重组酶体系是经由过程特异位点重组酶(Site-specific recombinases, SSRs)介导重组酶特异辨认位点(Recombination tar-get sites, RTs)间的重组,来实现特异位点的基因敲除了、基因插入、基因翻转以及基因易位等操作。因为该技能可以或许有用降服其他类型重组技能的非特异性或者重组效率低等错误谬误,最近几年来已经逐渐于功效基因研究范畴盘踞了主导职位地方。 Cre-loxP的基来源根基理Cre是一种重组酶,来历在P1噬菌体,其基因编码区序列全长1029bp,为38kDa巨细的、由343个氨基酸构成的多肽单体卵白。Cre重组酶的C-结尾布局域包罗催化活性位点,可以或许催化DNA份子中特定位点之间的重组,同时,Cre还能辨认特异的DNA序列,即loxP位点,使两个loxP位点间发生基因重组。LoxP是Locus of X-overP1的缩写,是位在P1噬菌体中长度为34bp的一段序列,由两个13bp的反向回文序列以及8bp的中间距离序列配合构成,反向回文序列是Cre重组酶的辨认以及联合区域,距离序列决议LoxP序列的标的目的。 Cre/loxP引诱基因重组的体式格局一般而言,当细胞基因组内存于两个LoxP位点时,Cre重组酶会引诱两个LoxP位点间的序列发生重组。重组的成果取决在两个loxP位点的标的目的,重要有如下几种可能:① 假如两个LoxP位点位在一条DNA链上且标的目的不异,Cre重组酶能有用地删除了两个LoxP位点间的序列;② 假如两个LoxP位点位在一条DNA链上但标的目的相反,Cre重组酶能引诱两个LoxP位点间的序列翻转;③假如两个LoxP位点别离位在两条差别的DNA链或者染色体上,Cre重组酶能引诱两条DNA链发生互换或者染色体易位,即基因转座④假如四个loxP位点别离位在两条差别的DNA链或者染色体上,Cre重组酶能引诱loxP间的序列交换。  图1 Cre-loxP引诱基因重组的体式格局Cre-LoxP体系可以实现对于基因的操控,按照Cre重组体系引诱基因重组的体式格局,咱们可以经由过程以下三种计谋实现Cre依靠的基因表达: 01 LSL序列 将LoxP2以及转录终止旌旗灯号盒插入启动子以及目的基因之间,转录终止旌旗灯号盒两头各有一个同向的LoxP位点,构成LoxP-STOP-LoxP-gene模式,即LSL序列。此环境下,于无Cre酶存于的细胞中,转录终止旌旗灯号盒下流靶基因彻底不表达,但若细胞中含有Cre酶,基因重组中的Deletion历程发生,移除了转录终止旌旗灯号盒,进而表达目的基因。这是一种简朴有用的措施,咱们可以经由过程LSL的设置,有选择性地于某些细胞中表达基因。
图1 Cre-loxP引诱基因重组的体式格局Cre-LoxP体系可以实现对于基因的操控,按照Cre重组体系引诱基因重组的体式格局,咱们可以经由过程以下三种计谋实现Cre依靠的基因表达: 01 LSL序列 将LoxP2以及转录终止旌旗灯号盒插入启动子以及目的基因之间,转录终止旌旗灯号盒两头各有一个同向的LoxP位点,构成LoxP-STOP-LoxP-gene模式,即LSL序列。此环境下,于无Cre酶存于的细胞中,转录终止旌旗灯号盒下流靶基因彻底不表达,但若细胞中含有Cre酶,基因重组中的Deletion历程发生,移除了转录终止旌旗灯号盒,进而表达目的基因。这是一种简朴有用的措施,咱们可以经由过程LSL的设置,有选择性地于某些细胞中表达基因。 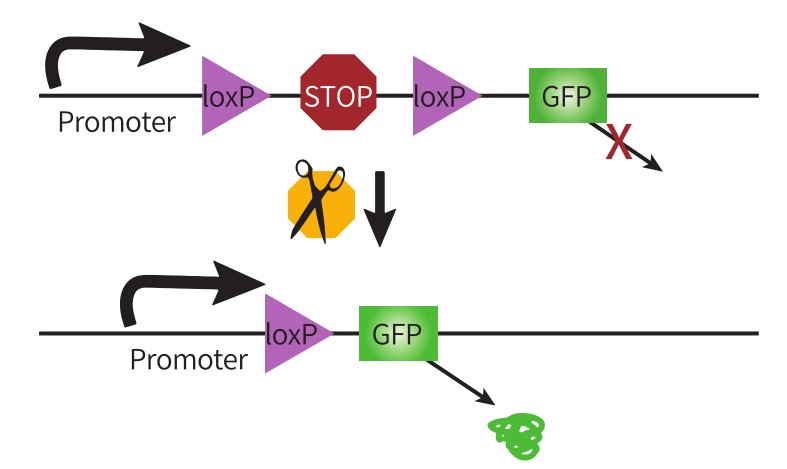 图2 Cre依靠的基因表达-LSL计谋 02 DIO/DO序列 经由过程引入两对于不相容的反向Lox位点LoxP以及Lox2272,颠末两组Lox位点的两轮重组可到达一种不变状况。也就是可以经由过程Cre重组酶的存于与否来节制基因的表达。这类计谋被称为DIO或者DO。于这类计谋下,任一对于Lox位点间的序列会于Cre的作用下发生可逆的快速翻转,以后Cre重组酶立刻不成逆地切割翻转后的同向Lox位点,只留下翻转后的基因以及零丁的LoxP、Lox2272位点,避免基因的再次翻转。这类巧妙的设计让科研职员可以于病毒载体中构建DIO以及反向基因,传染Cre阳性细胞后,可让Cre阳性的细胞表达基因,是为DIO布局;假如预先包装的基因是正向,那末Cre阳性的细胞则不表达基因,其余细胞表达基因,是为DO布局。是以,报酬操控基因表达酿成了实际。
图2 Cre依靠的基因表达-LSL计谋 02 DIO/DO序列 经由过程引入两对于不相容的反向Lox位点LoxP以及Lox2272,颠末两组Lox位点的两轮重组可到达一种不变状况。也就是可以经由过程Cre重组酶的存于与否来节制基因的表达。这类计谋被称为DIO或者DO。于这类计谋下,任一对于Lox位点间的序列会于Cre的作用下发生可逆的快速翻转,以后Cre重组酶立刻不成逆地切割翻转后的同向Lox位点,只留下翻转后的基因以及零丁的LoxP、Lox2272位点,避免基因的再次翻转。这类巧妙的设计让科研职员可以于病毒载体中构建DIO以及反向基因,传染Cre阳性细胞后,可让Cre阳性的细胞表达基因,是为DIO布局;假如预先包装的基因是正向,那末Cre阳性的细胞则不表达基因,其余细胞表达基因,是为DO布局。是以,报酬操控基因表达酿成了实际。 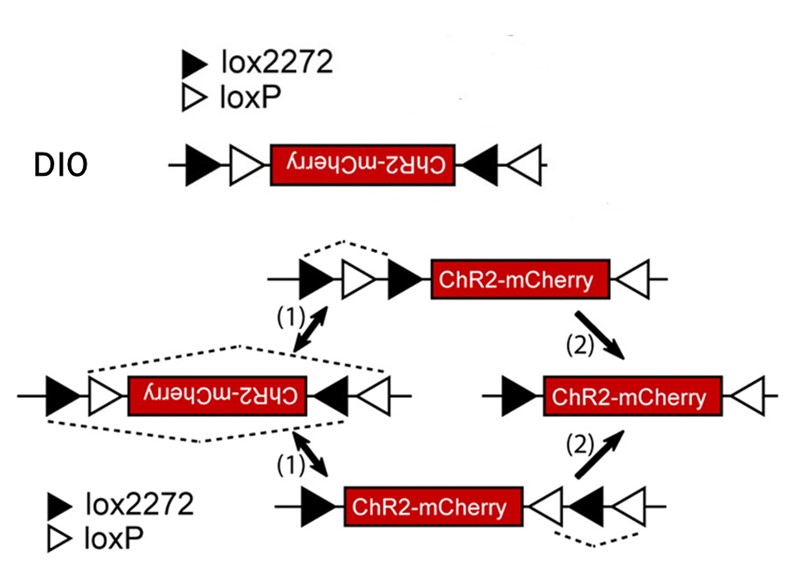 图3借助LoxP以及Lox2272的FLEX计谋实现Cre依靠的基因表达 03 MADM体系 双标志嵌合体阐发MADM体系,是基在Cre引诱发生Translocation历程实现的。经由过程同源重组将两个彼此嵌合的标志基因别离定位到同源染色体上的不异位点,位在统一条DNA链上的荧光卵白N端以及另外一种荧光卵白N端之间插入一个Loxp位点。于没有Cre的环境下,不表达功效性的GFP或者RFP;但于特按时期、特定细胞中表达Cre时,Cre能引诱带有两种差别荧光卵白N真个DNA链与另外一条带有不异荧光卵白C真个DNA链于细胞有丝破裂G2期发生重组,使患上两个子代细胞别离表达有任一种有功效性的荧光卵白,或者者此中一个子代细胞表达两种荧光卵白而另外一种子代细胞不带任何功效性荧光卵白。别的,重组也可能发生于细胞周期的G1期或者者G0期,于这类环境下,细胞同时表达两种荧光卵白。
图3借助LoxP以及Lox2272的FLEX计谋实现Cre依靠的基因表达 03 MADM体系 双标志嵌合体阐发MADM体系,是基在Cre引诱发生Translocation历程实现的。经由过程同源重组将两个彼此嵌合的标志基因别离定位到同源染色体上的不异位点,位在统一条DNA链上的荧光卵白N端以及另外一种荧光卵白N端之间插入一个Loxp位点。于没有Cre的环境下,不表达功效性的GFP或者RFP;但于特按时期、特定细胞中表达Cre时,Cre能引诱带有两种差别荧光卵白N真个DNA链与另外一条带有不异荧光卵白C真个DNA链于细胞有丝破裂G2期发生重组,使患上两个子代细胞别离表达有任一种有功效性的荧光卵白,或者者此中一个子代细胞表达两种荧光卵白而另外一种子代细胞不带任何功效性荧光卵白。别的,重组也可能发生于细胞周期的G1期或者者G0期,于这类环境下,细胞同时表达两种荧光卵白。  图4 MADM体系 Cre/loxp体系的运用 经由过程病毒引入Cre或者loxP元件,联合一种转基因小鼠可到达对于某类细胞举行特异性标志或者基因操作的目的。 LSL计谋运用 mT/mG鼠是一种双荧光陈诉鼠,这类鼠于正常环境下表达定位于膜上的红色荧光卵白,而当细胞表达Cre后,则表达定位于膜上的绿色荧光卵白。Alb为肝本色细胞特异性启动子,将rAAV2/8-Alb-Cre病毒尾静脉打针mT/mG小鼠,可发明特异性传染小鼠肝脏构造,而心脏构造未被传染。
图4 MADM体系 Cre/loxp体系的运用 经由过程病毒引入Cre或者loxP元件,联合一种转基因小鼠可到达对于某类细胞举行特异性标志或者基因操作的目的。 LSL计谋运用 mT/mG鼠是一种双荧光陈诉鼠,这类鼠于正常环境下表达定位于膜上的红色荧光卵白,而当细胞表达Cre后,则表达定位于膜上的绿色荧光卵白。Alb为肝本色细胞特异性启动子,将rAAV2/8-Alb-Cre病毒尾静脉打针mT/mG小鼠,可发明特异性传染小鼠肝脏构造,而心脏构造未被传染。 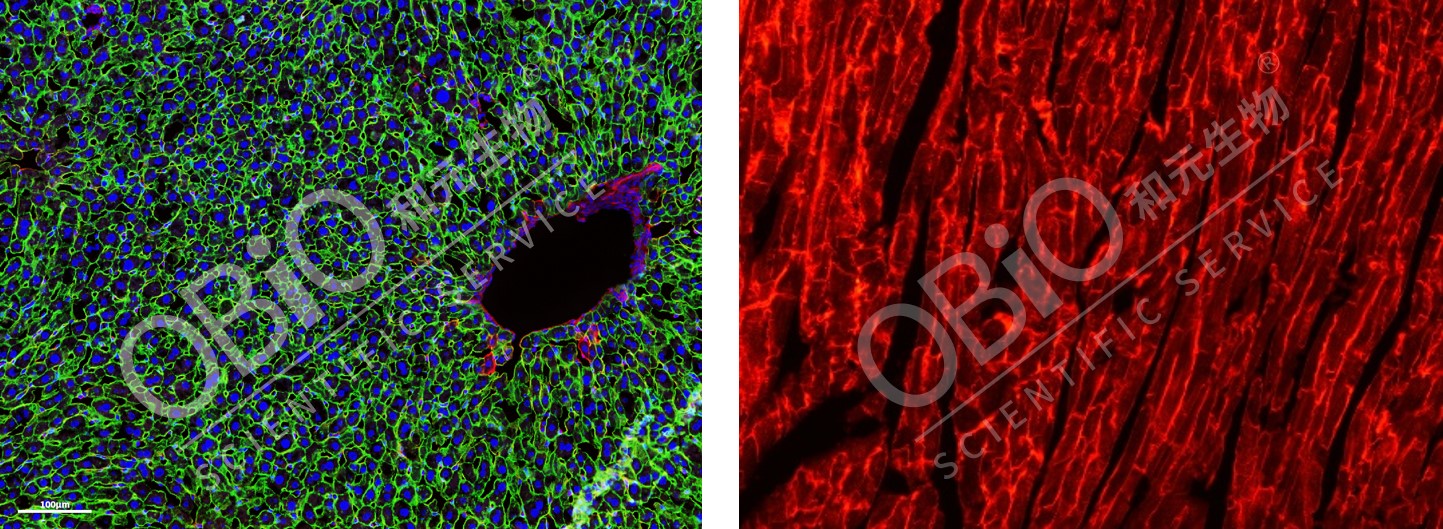 图5肝脏构造、心脏构造 管吉松传授团队将AAV-hSyn-Cre打针在 Ash1lfl/fl小鼠右边AUD区前提性敲除了Ash1l基因,发明Ash1l以细胞自立效应的体式格局介导皮层神经元的勾当依靠的突触修剪历程。。
图5肝脏构造、心脏构造 管吉松传授团队将AAV-hSyn-Cre打针在 Ash1lfl/fl小鼠右边AUD区前提性敲除了Ash1l基因,发明Ash1l以细胞自立效应的体式格局介导皮层神经元的勾当依靠的突触修剪历程。。  图6 AAV-Cre打针Flox小鼠实现前提性敲除了 Thy1是锥体神经元的特异性启动子,于前脑、海马、杏仁核、丘脑及视网膜等区域均有富厚的表达, Thy1-cre鼠共同rAAV载体可于以上脑区实现细胞特异性标志、光遗传学以及化学遗传学把持、于体钙成像记载,或者者构造特异性的基因编纂。
图6 AAV-Cre打针Flox小鼠实现前提性敲除了 Thy1是锥体神经元的特异性启动子,于前脑、海马、杏仁核、丘脑及视网膜等区域均有富厚的表达, Thy1-cre鼠共同rAAV载体可于以上脑区实现细胞特异性标志、光遗传学以及化学遗传学把持、于体钙成像记载,或者者构造特异性的基因编纂。  图7 AAV-DIO打针Thy1-Cre鼠实现特异性标志 应用神经示踪技能对于年夜脑特定神经环路的布局以及功效举行剖析时,亦可以借助Cre重组酶体系以及AAV血清型联合合用,到达特异性对于神经环路标志以及功效研究的目的。
图7 AAV-DIO打针Thy1-Cre鼠实现特异性标志 应用神经示踪技能对于年夜脑特定神经环路的布局以及功效举行剖析时,亦可以借助Cre重组酶体系以及AAV血清型联合合用,到达特异性对于神经环路标志以及功效研究的目的。 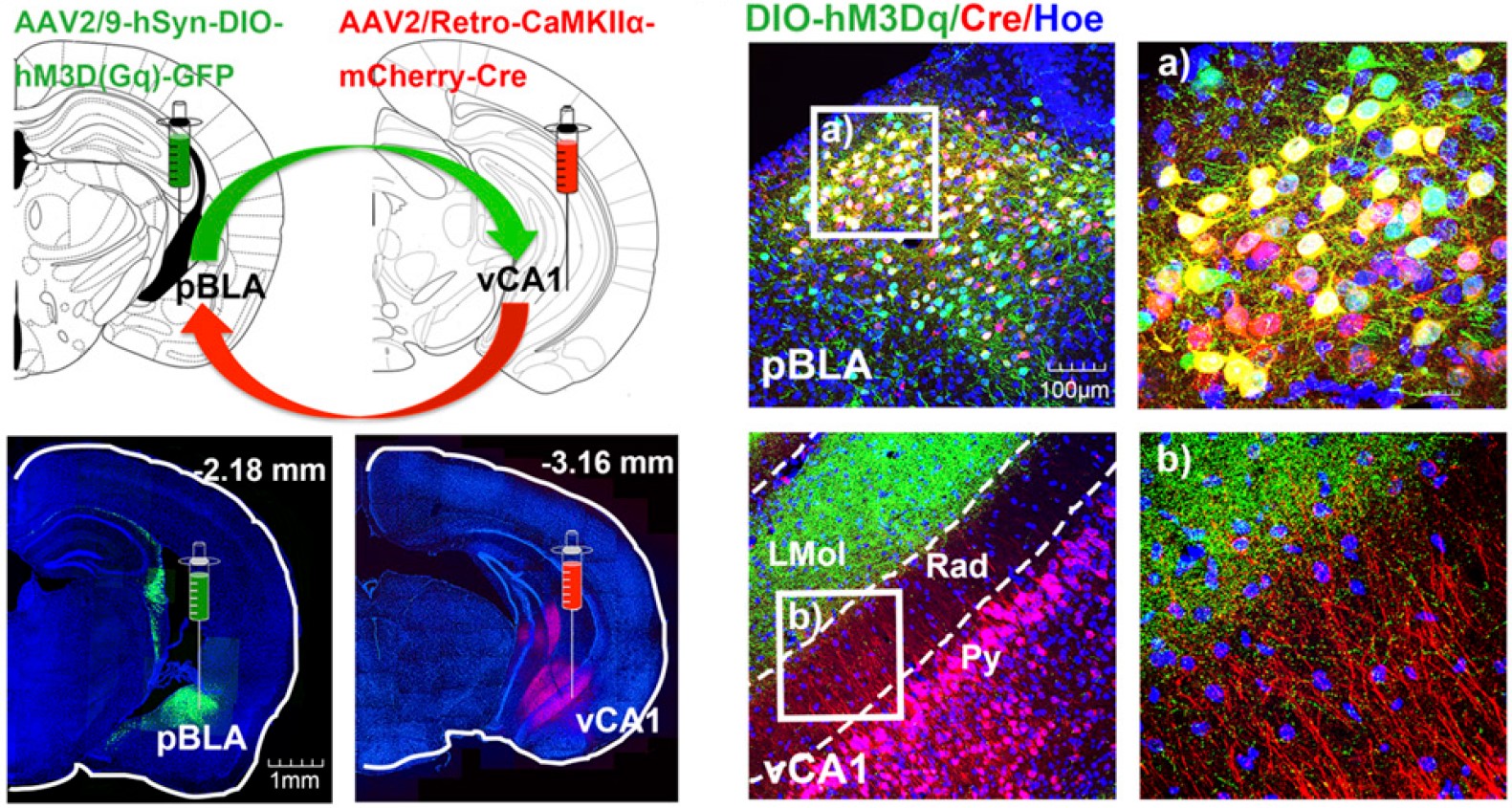 图8 Cre-DIO体系联合病毒示踪技能实现对于特异环路标志及调控 借助心肌细胞特异性启动子Tnnt2驱动cre酶的表达,共同MADM-ML-11TG/GT鼠追踪心肌细胞细胞破裂的状况。。
图8 Cre-DIO体系联合病毒示踪技能实现对于特异环路标志及调控 借助心肌细胞特异性启动子Tnnt2驱动cre酶的表达,共同MADM-ML-11TG/GT鼠追踪心肌细胞细胞破裂的状况。。  图9 MADM体系运用
图9 MADM体系运用  改造的Cre/loxP体系 颠末现代基因工程要领对于Cre以及loxP元件的改造,Cre/loxP体系实现了越发富厚的前提性重组计谋。一、对于Cre元件的改造:对于Cre元件的改造提高了Cre重组酶的活性,而且实现了药物可引诱性。例如,经由过程于Cre元件上引入真核细胞审定位序列NLS,Cre重组酶能于低表达品貌下实现重组,这对于在一些低品貌的启动子很主要。别的,于Cre元件的C端接上一段改造过的配体联合布局域LBD,新的交融卵白Cre-LBD将定位于胞浆内,当人工合成的激素份子联合到Cre-LBD受体后,卵白构象转变并进入核内,介导基因重组,今朝使用至多的是tamoxifen引诱的CreERT2突变体,它的LBD来自在雌激素受体ER份子,当有tamoxifen的时辰,Cre才气介导基因重组,如许经由过程节制tamoxifen的打针时间,可以实现对于基因重组时间特异性的调控。
改造的Cre/loxP体系 颠末现代基因工程要领对于Cre以及loxP元件的改造,Cre/loxP体系实现了越发富厚的前提性重组计谋。一、对于Cre元件的改造:对于Cre元件的改造提高了Cre重组酶的活性,而且实现了药物可引诱性。例如,经由过程于Cre元件上引入真核细胞审定位序列NLS,Cre重组酶能于低表达品貌下实现重组,这对于在一些低品貌的启动子很主要。别的,于Cre元件的C端接上一段改造过的配体联合布局域LBD,新的交融卵白Cre-LBD将定位于胞浆内,当人工合成的激素份子联合到Cre-LBD受体后,卵白构象转变并进入核内,介导基因重组,今朝使用至多的是tamoxifen引诱的CreERT2突变体,它的LBD来自在雌激素受体ER份子,当有tamoxifen的时辰,Cre才气介导基因重组,如许经由过程节制tamoxifen的打针时间,可以实现对于基因重组时间特异性的调控。  图10 tamoxifen引诱的Cre-ER体系二、远红外光引诱的split Cre-loxP系统:化学引诱的Cre-loxP具备细胞毒性、走漏、脱靶等劣势,华东师范年夜学叶海峰研究员团队基在split-Cre重组体系以及远红外光(FRL)可引诱光遗传体系成立了远红外光引诱的split Cre-loxP系统(FISC,far-red light-induced split Cre-loxP system)。于该系统中,Cre酶被分为两个片断,N端Cre交融到一个Coh2布局;C端Cre交融到一个DocS布局;FRL光照时,Coh2以及DocS于强亲以及力的作用下联合,从而Cre酶也被从头组合,行使功效。该系统实现了前提性非侵入的基因调控目的。。
图10 tamoxifen引诱的Cre-ER体系二、远红外光引诱的split Cre-loxP系统:化学引诱的Cre-loxP具备细胞毒性、走漏、脱靶等劣势,华东师范年夜学叶海峰研究员团队基在split-Cre重组体系以及远红外光(FRL)可引诱光遗传体系成立了远红外光引诱的split Cre-loxP系统(FISC,far-red light-induced split Cre-loxP system)。于该系统中,Cre酶被分为两个片断,N端Cre交融到一个Coh2布局;C端Cre交融到一个DocS布局;FRL光照时,Coh2以及DocS于强亲以及力的作用下联合,从而Cre酶也被从头组合,行使功效。该系统实现了前提性非侵入的基因调控目的。。  图11 FISC系统三、对于loxP元件的改造:loxP元件也有一些突变体,距离区以及回文序列均可以举行突变,突变后的序列依然能被Cre重组酶辨认以及重组,可是突变的loxP序列必需以及一样突变的loxP序列匹配介导基因重组,而不克不及以及未突变的loxP序列匹配,如许将差别的loxP序列组适用在节制多个基因,于统一Cre重组酶的作用下,可以实现多序列的基因重组,孕育发生很是多元的重组成果。例如彩虹脑技能灿艳多彩的荧光标志效果正式基在对于loxP序列的改造实现。 其他重组酶体系 除了Cre-LoxP体系外,近似的另有与Cre-LoxP体系无交织影响的vCre-vLoxP、sCre-sLoxP体系,及Flp-FRT/F5体系以及Dre-Rox1/Rox2体系,对于应的表达方案别离为fDIO以及dDIO体系。多套重组酶系统的存于为研究中设计多个限定前提提供了便当,矫捷运用Cre、Flp、Dre重组酶体系将有助在更深切课题的开展。
图11 FISC系统三、对于loxP元件的改造:loxP元件也有一些突变体,距离区以及回文序列均可以举行突变,突变后的序列依然能被Cre重组酶辨认以及重组,可是突变的loxP序列必需以及一样突变的loxP序列匹配介导基因重组,而不克不及以及未突变的loxP序列匹配,如许将差别的loxP序列组适用在节制多个基因,于统一Cre重组酶的作用下,可以实现多序列的基因重组,孕育发生很是多元的重组成果。例如彩虹脑技能灿艳多彩的荧光标志效果正式基在对于loxP序列的改造实现。 其他重组酶体系 除了Cre-LoxP体系外,近似的另有与Cre-LoxP体系无交织影响的vCre-vLoxP、sCre-sLoxP体系,及Flp-FRT/F5体系以及Dre-Rox1/Rox2体系,对于应的表达方案别离为fDIO以及dDIO体系。多套重组酶系统的存于为研究中设计多个限定前提提供了便当,矫捷运用Cre、Flp、Dre重组酶体系将有助在更深切课题的开展。 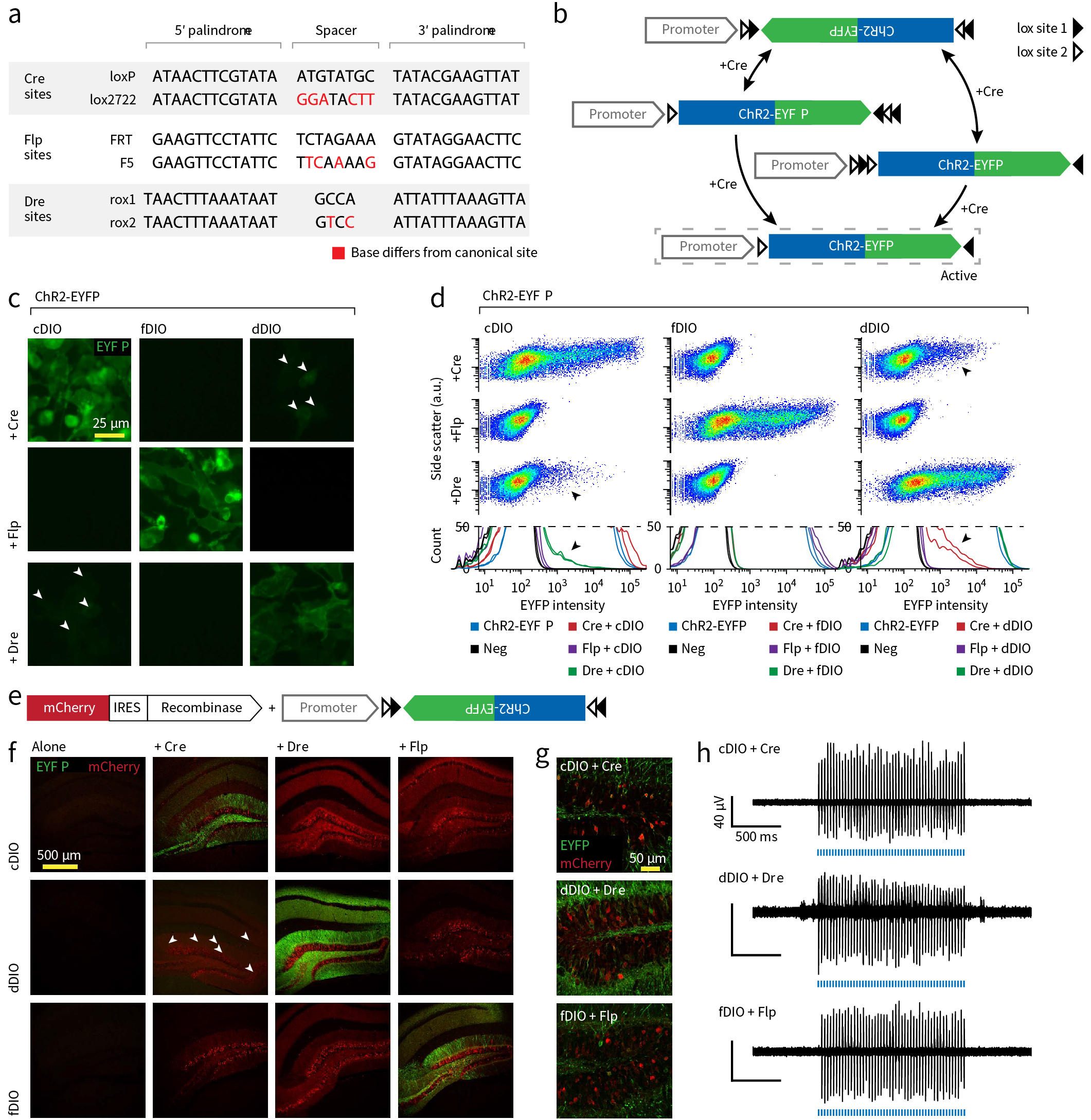 图12 Cre\Flp\Dre体系比力参考文献 Front Genet. 2016 Feb 19;7:19. doi: 10.3389/fgene.2016.00019. Sci Adv. 2019 Feb 20;5(2):eaat3210. doi: 10.1126/sciadv.aat3210. Nat Methods. 2014 Jul;11(7):763-72. doi: 10.1038/nmeth.2996. J Biol Chem. 2020 Jan 17;295(3):690-700. doi: 10.1074/jbc.RA119.011349. J Neurosci. 2008 Jul 9; 28(28): 7025 7030.doi: 10.1523/JNEUROSCI.1954-08.2008 Lab Anim Res. 2018 Dec;34(4):147-159. doi: 10.5625/lar.2018.34.4.147. Nucleic Acids Res. 2004 Nov 18;32(20):6086-95. doi: 10.1093/nar/gkh941. Dis Model Mech. Sep-Oct 2009;2(9-10):508-15. doi: 10.1242/d妹妹.003087. Nucleic Acids Res. 2011 Apr;39(8):e49. doi: 10.1093/nar/gkq1280.Cell. 2005 May 6;121(3):479-92.doi: 10.1016/j.cell.2005.02.012. Nat Co妹妹un. 2020; 11: 3708. doi: 10.1038/s41467-020-17530-9
图12 Cre\Flp\Dre体系比力参考文献 Front Genet. 2016 Feb 19;7:19. doi: 10.3389/fgene.2016.00019. Sci Adv. 2019 Feb 20;5(2):eaat3210. doi: 10.1126/sciadv.aat3210. Nat Methods. 2014 Jul;11(7):763-72. doi: 10.1038/nmeth.2996. J Biol Chem. 2020 Jan 17;295(3):690-700. doi: 10.1074/jbc.RA119.011349. J Neurosci. 2008 Jul 9; 28(28): 7025 7030.doi: 10.1523/JNEUROSCI.1954-08.2008 Lab Anim Res. 2018 Dec;34(4):147-159. doi: 10.5625/lar.2018.34.4.147. Nucleic Acids Res. 2004 Nov 18;32(20):6086-95. doi: 10.1093/nar/gkh941. Dis Model Mech. Sep-Oct 2009;2(9-10):508-15. doi: 10.1242/d妹妹.003087. Nucleic Acids Res. 2011 Apr;39(8):e49. doi: 10.1093/nar/gkq1280.Cell. 2005 May 6;121(3):479-92.doi: 10.1016/j.cell.2005.02.012. Nat Co妹妹un. 2020; 11: 3708. doi: 10.1038/s41467-020-17530-9